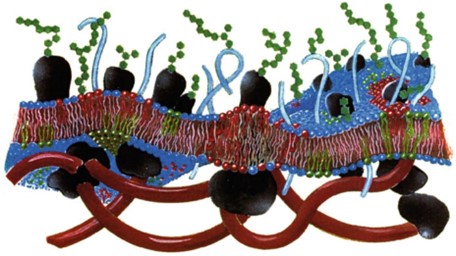
Retos en el estudio de membranas biológicas usando simulaciones de dinámica molecular
DOI:
https://doi.org/10.36790/epistemus.v19i38.357Palabras clave:
lípidos, bicapa, simulaciones, membrana, fasesResumen
En el estudio de simulaciones de membrana modelo, la selección de componentes resulta crucial. Incluso al considerar solo la bicapa, núcleo esencial de la membrana, distintos lípidos generan variadas interacciones, influyendo en el comportamiento del sistema y dando lugar a diversos procesos biológicos. Considerar escalas temporales adecuadas es esencial, ya que distintos fenómenos biológicos operan en intervalos temporales específicos. La elección de una escala inapropiada podría pasar por alto detalles intrínsecos al fenómeno en estudio.
En el presente estudio exploramos dos escalas de simulación: la resolución detallada de todos los átomos y la simplificación de grano grueso, destacando su impacto en precisión y rendimiento computacional. Abordamos la complejidad estudiando componentes individuales, como la bicapa. Este enfoque proporciona una perspectiva valiosa para comprender procesos biológicos complejos en la membrana plasmática, resaltando la importancia de elecciones cuidadosas en la simulación de sistemas biomiméticos.
Descargas
Citas
M. Feig, G. Nawrocki, I. Yu, “Challenges and opportunities in connecting simulations with experiment via molecular dynamics of cellular environments”, J. Phys. Conf. Ser, vol. 1036, no. 1, Enero 2018, DOI: http://10.1088/1742-6596/1036/1/012010. DOI: https://doi.org/10.1088/1742-6596/1036/1/012010
J. D. Murray, J. J. Lange, H. Bennett-Lenane, R. Holm, “Advancing algorithmic drug product development: Recommendations for machine learning approaches in drug formulation”, Eur. J. Pharm. Sci., vol. 191, Diciembre 2023, DOI: http://10.1016/j.ejps.2023.106562. DOI: https://doi.org/10.1016/j.ejps.2023.106562
I. Vattulainen, T. Rog, “Lipid simulations: A perspective on lipids in action”, Cold Spring Harb. Perspect. Biol., vol. 3, no. 4, Abril 2011, DOI: http://10.1101/cshperspect.a004655 . DOI: https://doi.org/10.1101/cshperspect.a004655
T. Heimburg, R. L. Biltonen, “A Monte Carlo simulation study of protein-induced heat capacity changes and lipid-induced protein clustering”, Biophys. J., vol. 70, pp. 84-96, Enero 1996, DOI: http://10.1016/S0006-3495(96)79551-6. DOI: https://doi.org/10.1016/S0006-3495(96)79551-6
R. X . Gu, S. Baoukina, D. P. Tieleman, “Phase separation in atomistic simulations of model membranes” J. Am. Chem. Soc., vol. 142, no. 6, pp. 2844-2856, Enero 2020, DOI: http://10.1021/jacs.9b11057. DOI: https://doi.org/10.1021/jacs.9b11057
L. Karami, “Interactions of neutral and protonated Tamoxifen with the DPPC lipid bilayer using molecular dynamics simulation”, Steroids, vol. 194, Enero 2023, DOI: http://10.1016/j.steroids.2023.109225. DOI: https://doi.org/10.1016/j.steroids.2023.109225
H. I. Ingólfsson, M. N. Melo, “Lipid Organization of the plasma membrane”, Journal of the American Chemical Society, vol.136, no. 41, pp. 14554-14559, Septiembre 2014, DOI: http://10.1021/ja507832e. DOI: https://doi.org/10.1021/ja507832e
E. Sezgin, “Giant plasma membrane vesicles to study plasma membrane structure and dynamics”, Biochim. Biophys. Acta. Biomembr., vol. 1864, no. 4, Abril 2022, DOI: http://10.1016/j.bbamem.2021.183857. DOI: https://doi.org/10.1016/j.bbamem.2021.183857
J. D. Nickels, “Lipid Rafts: Buffers of Cell Membrane Physical Properties”, J. Phys. Chem B, vol. 123, pp. 2050-2056, Enero 2019, DOI: https://doi.org/10.1021/acs.jpcb.8b12126. DOI: https://doi.org/10.1021/acs.jpcb.8b12126
E. Sezgin, I. Levental, S. Mayor, C. Eggeling, “The mystery of membrane organization: Composition, regulation and roles of lipid rafts”, Nature Reviews Molecular Cell Biology, vol. 18, no. 6, pp. 361–374, Junio 2017, DOI: https://doi.org/10.1038/nrm.2017.16. DOI: https://doi.org/10.1038/nrm.2017.16
S. L. Veacht, N. Rogers, A. Decker, S. A. Shelby, “The plasma membranes as an adaptable fluid mosaic”, Biochim. Biophys. Acta Biomembr., vol. 1865, no. 3, Marzo 2023, DOI: https://doi.org/10.1016/j.bbamem.2022.184114. DOI: https://doi.org/10.1016/j.bbamem.2022.184114
M. Doktorova, G. Khelashvili, R. Ashkar, M. F. Brown, “Molecular simulations and NMR reveal how lipid fluctuations affect membrane mechanics”, Biophys. J., vol. 122, no. 6, pp. 984-1002, Diciembre 2022, DOI: https://doi.org/10.1016/j.bpj.2022.12.007. DOI: https://doi.org/10.1016/j.bpj.2022.12.007
S. Qian, V. K. Sharma, L. A. Clifton, “Understanding the structure and dynamics of complex biomembrane interactions by neutron scattering techniques”, Langmuir, vol. 36, pp. 15189-15211, Diciembre 2020, DOI: https://doi.org/10.1021/acs.langmuir.0c02516. DOI: https://doi.org/10.1021/acs.langmuir.0c02516
R. Metzler, J. H. Jeon, A.G. Cherstvy, “Non-brownian diffusion in lipid membranes: experiments and simulations”, Biochim. Biophys. Acta. Biomembr., vol. 1858, no. 10, pp. 2451-2467, Octubre 2016, DOI: https://doi.org/10.1016/j.bbamem.2016.01.022. DOI: https://doi.org/10.1016/j.bbamem.2016.01.022
L. Porcar, Y. Gerelli, “On the lipid flip-flop and phase transition coupling”, Soft Matter, vol. 16, no. 33, pp. 7696-7703, Agosto 2020, DOI: https://doi.org/10.1039/D0SM01161D. DOI: https://doi.org/10.1039/D0SM01161D
E. L. Wu, X. Cheng, S. Jo, H. Rui, K. C. Song, “CHARMM-GUI membrane builder toward realistic biological membrane simulations”, J. Comput. Chem., vol.35, no. 27, pp. 1997-2004, Octubre 2014, DOI: https://doi.org/10.1002/jcc.23702. DOI: https://doi.org/10.1002/jcc.23702
S. J. Marrink, H. J. Risselada, S. Yefimov, D. P. Tieleman, “The MARTINI force field: Coarse grained model for biomolecular simulations”, J. Phys. Chem. B., vol. 111, pp. 7812-7824, Junio 2007, DOI: https://doi.org/10.1021/jp071097f. DOI: https://doi.org/10.1021/jp071097f
H. Rakhshani, E. Dehghanian, A. Rahati, “Enhanced GROMACS: toward a better numerical simulation framework”, J. Mol. Model, vol. 25, no. 12, Noviembre 2019, DOI: https://doi.org/10.1007/s00894-019-4232-z. DOI: https://doi.org/10.1007/s00894-019-4232-z
P. Van Der Ploeg, H. J. C. Berendsen, “Molecular dynamics of a bilayer membrane”, J. Chem. Phys, vol. 76, no. 6, pp. 3271-3276, Marzo 1982, DOI: https://doi.org/10.1007/978-1-62703-017-5_15. DOI: https://doi.org/10.1063/1.443321
S. J. Marrink, V. Corradi, H. I. Ingólfsson, D. P. Tieleman, “Computational Modelling of realistic cell membranes”, Chem. Rev., vol. 119, no. 9, pp. 6184-6226, Mayo 2019, DOI: https://doi.org/10.1021/acs.chemrev.8b00460. DOI: https://doi.org/10.1021/acs.chemrev.8b00460
J. Risbo, M. M. Sperotto, O. G. Mouritsen, “Theory of phase equilibria and critical mixing points in binary lipid bilayers”, J. Chem. Phys, vol. 103, no. 9, pp. 3643-3656, Mayo 1995, DOI: https://doi.org/10.1063/1.470041. DOI: https://doi.org/10.1063/1.470041
O. G. Mouritsen, L. A. Bagatolli, Life - as a matter of fat lipids in a membrane biophysics perspective. Segunda edición. Suiza: Springer International Publishing Switzerland, 2005, DOI: https://doi.org/10.13140/RG.2.1.1190.4723.
D. Marsh, “Cholesterol-induced fluid membrane domains: A compendium of lipid-raft ternary phase diagrams”, Biochimica et Biophysica Acta – Biomembranes, vol. 1788, no. 10, pp. 2114–2123, Agosto 2009, DOI: https://doi.org/10.1016/j.bbamem.2009.08.004. DOI: https://doi.org/10.1016/j.bbamem.2009.08.004
I. Levental, K. R. Levental, F. A. Heberle, “Lipid Rafts: Controversies Resolved, Mysteries Remain”, Trends in Cell Biology, vol. 30, no. 5, pp. 341–353, Mayo 2020, DOI: https://doi.org/10.1016/j.tcb.2020.01.009. DOI: https://doi.org/10.1016/j.tcb.2020.01.009
K. Simons, E. Ikonen, “Functional rafts in cell membranes”, Nature, vol. 387, pp. 569–572, Junio 1997, DOI: https://doi.org/10.1038/42408. DOI: https://doi.org/10.1038/42408
I. Levental, S. L. Veatch, “The Continuing Mystery of Lipid Rafts”, Journal of Molecular Biology, vol. 428, no. 24, pp. 4749–4764, Agosto 2016, DOI: https://doi.org/10.1016/j.jmb.2016.08.022. DOI: https://doi.org/10.1016/j.jmb.2016.08.022
A. D. Maldonado Arce, C. Contreras Aburto, F. F. Rosales, J. A. Arvayo Zatarain, E. Urrutia Bañuelos, “Métodos de simulación computacional en biología”, EPISTEMUS, no. 21, 84-92, Diciembre 2016, DOI: https://doi.org/10.36790/epistemus.v10i21.38. DOI: https://doi.org/10.36790/epistemus.v10i21.38
M. P. Allen, D. J. Tildesley, Computer Simulation of Liquids, 2nd. Ed., Oxford University Press, 2017, DOI: https://doi.org/10.1093/oso/9780198803195.001.0001. DOI: https://doi.org/10.1093/oso/9780198803195.001.0001
D. Frenkel, B. Smit, Understanding Molecular Simulation From Algorithms to Applications, 2nd. Ed., Academic Press, 2002, DOI: https://doi.org/10.1063/1.881812. DOI: https://doi.org/10.1063/1.881812
M. Parrinello, A. Rahman, “Polymorphic transitions in single crystals: A new molecular dynamics method”, J. Appl. Phys., vol. 52, no. 12, pp. 7182-7190, Diciembre 1981, DOI: https://doi.org/10.1063/1.328693. DOI: https://doi.org/10.1063/1.328693
S. Nose, “A unified formulation of the constant temperature molecular dynamics methods”, J. Phys. Chem. Lett., vol. 81, no. 1, pp. 511-519, Julio 1984, DOI: https://doi.org/10.1063/1.447334. DOI: https://doi.org/10.1063/1.447334
R. W. Pastor, A. D. MacKerell, “Development of the CHARMM force field for lipids”, J. Phys. Chem. Lett., vol. 2, no. 13, pp. 1526-1532, Junio 2011, DOI: https://doi.org/10.1021/jz200167q. DOI: https://doi.org/10.1021/jz200167q
P. S. Nerenberg, T. Head-Gordon, “New developments in force fields for biomolecular simulations”, Current Opinion in Structural Biology, vol. 49, pp. 129-138, Abril 2018, DOI: https://doi.org/10.1016/j.sbi.2018.02.002. DOI: https://doi.org/10.1016/j.sbi.2018.02.002
Publicado
Cómo citar
Número
Sección
Licencia
Derechos de autor 2025 EPISTEMUS

Esta obra está bajo una licencia internacional Creative Commons Atribución-NoComercial-CompartirIgual 4.0.
La revista adquiere los derechos patrimoniales de los artículos sólo para difusión sin ningún fin de lucro, sin menoscabo de los propios derechos de autoría.
Los autores son los legítimos titulares de los derechos de propiedad intelectual de sus respectivos artículos, y en tal calidad, al enviar sus textos expresan su deseo de colaborar con la Revista Epistemus, editada semestralmente por la Universidad de Sonora.
Por lo anterior, de manera libre, voluntaria y a título gratuito, una vez aceptado el artículo para su publicación, ceden sus derechos a la Universidad de Sonora para que la Universidad de Sonora edite, publique, distribuya y ponga a disposición a través de intranets, internet o CD dicha obra, sin limitación alguna de forma o tiempo, siempre y cuando sea sin fines de lucro y con la obligación expresa de respetar y mencionar el crédito que corresponde a los autores en cualquier utilización que se haga del mismo.
Queda entendido que esta autorización no es una cesión o transmisión de alguno de sus derechos patrimoniales en favor de la mencionada institución. La UniSon le garantiza el derecho de reproducir la contribución por cualquier medio en el cual usted sea el autor, sujeto a que se otorgue el crédito correspondiente a la publicación original de la contribución en Epistemus.
Salvo indicación contraria, todos los contenidos de la edición electrónica se distribuyen bajo una licencia de uso y Attribution-NonCommercial-ShareAlike 4.0 International (CC BY-NC-SA 4.0) Puede consultar desde aquí la versión informativa y el texto legal de la licencia. Esta circunstancia ha de hacerse constar expresamente de esta forma cuando sea necesario.